Inciensa secuencia el genoma completo del primer caso de virus de la viruela símica
Jueves 11 de agosto, 2022. A partir de la muestra recibida del primer caso de viruela símica, Inciensa consiguió realizar la secuenciación del mismo, siendo el primer país centroamericano en lograrlo.
La muestra se secuenció por la técnica de metagenómica en escopeta (del inglés shotgun metagenomics); esta metodología consistió en tomar la muestra del paciente, hacer una extracción de todo el material genético presente (ADN del paciente, del microbiota acompañante y del virus), secuenciarlo por la tecnología de secuenciación de siguiente generación y posteriormente, al realizar los análisis computacionales (bioinformáticos), se identificó el material genético del virus y se reconstruyó su genoma.
Una vez reconstruido el genoma viral completo se comparó contra los genomas reportados por los demás países donde se han presentado casos de viruela símica y se determinó a cuál clado o rama evolutiva pertenecía el virus.
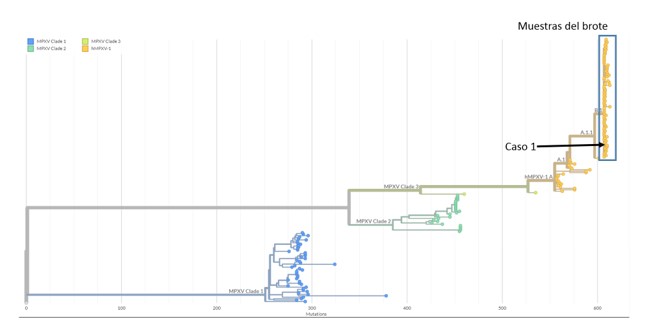
“La secuenciación de este virus es de gran importancia epidemiológica, ya que permite identificar el clado al cual pertenece el virus. Los clados se han asociado con diferentes grados de severidad de la enfermedad y tasas de mortalidad: para el clado 1, se reporta una letalidad de una de cada 10 personas infectadas en los países de África central. El clado 2 y 3 causa una enfermedad más leve y una tasa de mortalidad más baja (estimada en alrededor del 1%) y se ha descrito principalmente en África occidental”, agregó el Dr. Francisco Duarte coordinador del laboratorio de genómica y biología molecular del Inciensa.
La secuencia del primer caso detectado en Costa Rica según esta nomenclatura pertenece al clado 3, que corresponde al clado que está produciendo el reciente brote de la enfermedad a nivel mundial. La epidemiología genómica del virus de la viruela símica puede ser consultada en: https://nextstrain.org/monkeypox?c=country
El equipo multidisciplinario del laboratorio de genómica del Inciensa y sus colaboradores de la Universidad de Costa Rica (quienes han venido llevando a cabo la vigilancia genómica de SARS-CoV-2) seleccionaron los protocolos de laboratorio adecuados para este tema y además evaluaron las estrategias bioinformáticas para analizar los datos de la secuenciación. Estas estrategias permiten generar y contextualizar los resultados genómicos de los casos que se puedan secuenciar en Costa Rica e interpretarlos a la luz de la epidemiología genómica global para seguimiento del brote.
Desde el Centro Nacional de Referencia de Virología (CNRV) del Inciensa se continuará coordinando con la red de laboratorios tanto públicos (CCSS) como privados para identificar y diagnosticar oportunamente los posibles casos de esta enfermedad, con el objetivo de que el sistema de salud pueda aplicar las medidas de prevención y control adecuadas.